Introduction
Dans cette article, nous allons voir comment faire du clustering avec DBSCAN (density-based spatial clustering of applications with noise). D’abord, nous allons expliquer le clustering en répondant à ces deux questions : c’est quoi ? quels sont les critères qu’il doit satisfaire ? Ensuite, nous allons nous concentrer sur DBSCAN pour le decouvrir et voir son fonctionnement. Pour Terminer, nous ferons un petit TP sur DBSCAN.
Clustering
Définition
D’aprés wikipedia, Le clustering est une méthode en analyse des données. Elle vise à diviser un ensemble de données en différents « paquets » homogènes, en ce sens que les données de chaque sous-ensemble partagent des caractéristiques communes, qui correspondent le plus souvent à des critères de proximité (similarité informatique) que l’on définit en introduisant des mesures et classes de distance entre objets.
Les algorithmes de clustering font partie de la classe des algorithmes d’apprentissages non supervisés. Il existe différents algorithmes de clustering parmis lesquels on peut citer : le clustering hiérarchique, K-means, et DBSCAN. Dans cette Article nous n’allons aborder que DBSCAN.
Critères que doit satisfaire un clustering
Dans le cas d’algorithmes non supervisés, les criteres que doit satisfaire l’algorithme sont moins évident à définir que dans le cas d’algorithmes supervisés, où il y a une tâche claire à accomplir. Ceci n’empêche pas qu’il existe des mesures de la performance d’un algorithme non supervisé :
a – Distances et similarités
Faire un clustering, c’est regrouper ensemble les points les plus proches, ou les plus semblables. Le concept de clustering repose fortement sur ceux de distance et de similarité. Ces concepts sont très utiles pour définir à quel point deux éléments sont proches les unes des autres ; à quel point un élément est proche d’un cluster ; et à quel point deux clusters sont proches l’un de l’autre.
Les exemples les plus utilisés de distances sont la distance euclidienne et la distance de Manhattan.
b – Forme des clusters
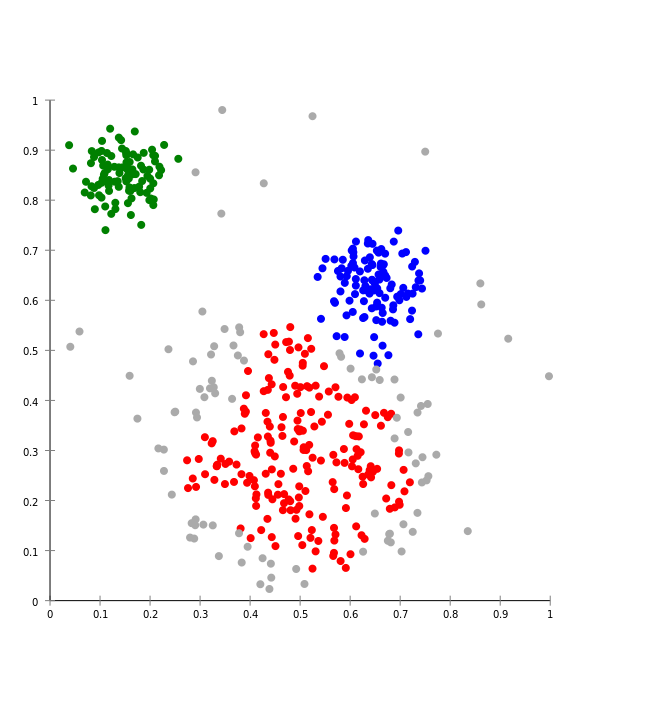
Nous voulons de nos clusters qu’ils soient
1. resserrés sur eux mêmes (deux points qui sont proches doivent appartenir au même cluster)
On appelle centroïde d’un cluster le barycentre des points de ce cluster :
\(µ_{k}=\frac{1}{|C_{k}|}\sum_{x∈C_{k}}x\)
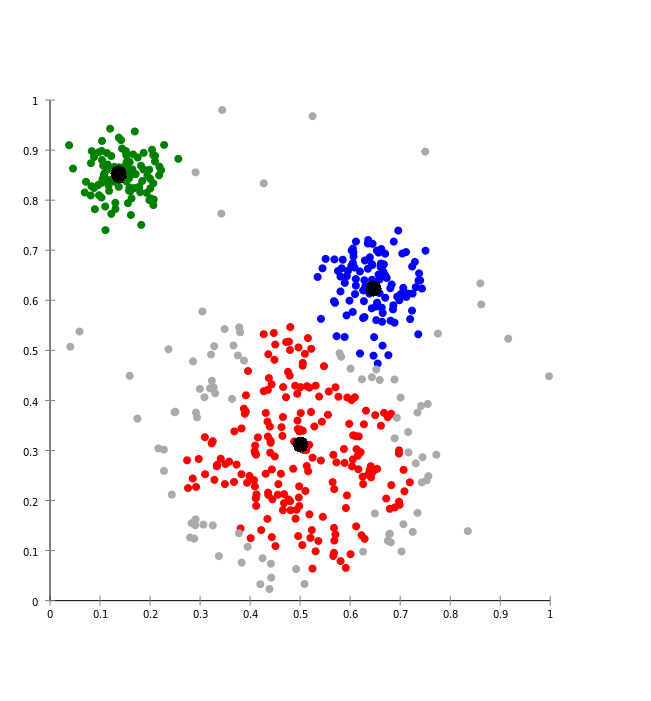
(crédit : image modifiée de Chire – CC BY-SA 3.0)
On définit ainsi l’homogénéité (notée Tk pour tightness en anglais) d’un cluster comme la moyenne des distances de chacun des points contenus dans ce cluster au centroïde μk:
\(T_{k}=\frac{1}{|C_{k}|}\sum_{x∈C_{k}}d(x,µ_{k})\)
Un cluster resserré aura une hétérogénéité plus faible qu’un cluster de points épars.
Pour caractériser non pas un cluster, mais l’ensemble des clusters, on peut calculer la moyenne des homogénéités de chaque cluster.
2. loin les uns des autres (deux points qui sont éloignés doivent appartenir à des clusters différents)
Pour quantifier le fait que deux clusters soient loin l’un de l’autre, on définit la séparation de deux clusters comme la distance entre leurs centroïdes :
\(S_{k,l}=d(µ_{k},µ_{l})\)
On peut calculer la moyenne de ces quantités sur l’ensemble des paires de cluster (k,l) obtenues :
\(S=\frac{2}{k(k-1)}\sum_{k=1}^K\sum_{l=k+1}^KS_{k,l}\)
c – Indice de Davies-Bouldin
Nous avons maintenant deux critères à optimiser : les distannce intra-cluster (c’est l’homogénéité), que l’on veut faibles, aux distances inter-cluster (la séparation), que l’on veut grandes. L’idée de cette indice est de comparer ces deux critères.
Pour un cluster k, cet indice est donné par :
\(D_{k}=max_{l≠k}\frac{T_{l}+T_{k}}{S_{k,l}}\)
cet indice est d’autant plus faible que tous les clusters sont homogènes (le numérateur de cette fraction est petit) et que tous sont bien séparés (le dénominateur est grand). On peut calculer cet indice de maniere global en moyennant les indices de Davies-Bouldin de tous les clusters.
d – coefficient de silhouette :
L’autre moyen de quantifier à quel point un clustering répond à ces deux exigences (homogénéité et séparation) est de mesurer le coefficient de silhouette.
Pour un point x donné, le coefficient de silhouette s(x) permet d’évaluer si ce point appartient au « bon » cluster :
– x est-il proche des points du cluster auquel il appartient ?
pour repondre à cette question, on calcule la distance moyenne de x à tous les autres points du cluster C_k auquel il appartient :
\(a(x)=\frac{1}{|C_{k}|-1}\sum_{u∈C_{k},u≠x}d(u,x)\)
– x Est-il loin des autres points ?
pour repondre à cette question, on calcule la plus petite valeur que pourrait prendre a(x), si x était assigné à un autre cluster :
\(b(x)=min_{l≠k}\frac{1}{|C_{l}|}\sum_{u∈C_{l}}d(u,x)\)
Le coefficient de silhouette est donné par :
\(s(x)=\frac{b(x)-a(x)}{max(a(x),b(x))}\)
DBSCAN
Définition
DBSCAN (density-based spatial clustering of applications with noise) est un algorithme de partitionnement de données proposé en 1996 par Martin Ester, Hans-Peter Kriegel, Jörg Sander et Xiaowei Xu1. Il s’agit d’un algorithme fondé sur la densité dans la mesure qui s’appuie sur la densité estimée des clusters pour effectuer le partitionnement.
Fonctionnement de DBSCAN
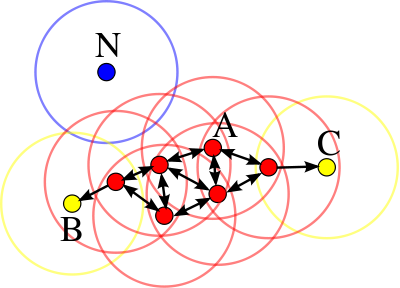
L’algorithme DBSCAN utilise 2 paramètres : la distance ε et le nombre minimum de points « MinPts » devant se trouver dans un rayon ε pour que ces points soient considérés comme un cluster.
DBSCAN fonction de la maniere suivante :
1 – DBSCAN commence par un point de données de départ arbitraire qui n’a pas été visité. Le voisinage de ce point est extrait en utilisant une distance epsilon ε.
2 – S’il y a un nombre suffisant de points (selon les minPoints) dans ce voisinage, le processus de mise en cluster démarre et le point de données actuel devient le premier point du nouveau cluster. Sinon, le point sera étiqueté comme bruit (plus tard, ce point bruyant pourrait devenir la partie du cluster). Dans les deux cas, ce point est marqué comme «visité».
3 – Pour ce premier point du nouveau cluster, les points situés dans son voisinage à distance se joignent également au même cluster. Cette procédure est ensuite répétée pour tous les nouveaux points qui viennent d’être ajoutés au groupe de cluster.
4 – Ce processus des étapes 2 et 3 est répété jusqu’à ce que tous les points du cluster soient déterminés, c’est-à-dire que tous les points à proximité du ε voisinage du cluster ont été visités et étiquetés.
5 – Une fois terminé avec le cluster actuel, un nouveau point non visité est récupéré et traité, ce qui permet de découvrir un nouveau cluster ou du bruit. Ce processus se répète jusqu’à ce que tous les points soient marqués comme étant visités. A la fin de tous les points visités, chaque points a été marqué comme appartenant à un cluster ou comme étant du bruit.
Algorithme
DBSCAN(D, eps, MinPts)
C = 0
pour chaque point P non visité des données D
marquer P comme visité
PtsVoisins = epsilonVoisinage(D, P, eps)
si tailleDe(PtsVoisins) < MinPts
marquer P comme BRUIT
sinon
C++
etendreCluster(D, P, PtsVoisins, C, eps, MinPts)
etendreCluster(D, P, PtsVoisins, C, eps, MinPts)
ajouter P au cluster C
pour chaque point P' de PtsVoisins
si P' n'a pas été visité
marquer P' comme visité
PtsVoisins' = epsilonVoisinage(D, P', eps)
si tailleDe(PtsVoisins') >= MinPts
PtsVoisins = PtsVoisins U PtsVoisins'
si P' n'est membre d'aucun cluster
ajouter P' au cluster C
epsilonVoisinage(D, P, eps)
retourner tous les points de D qui sont à une distance inférieure à epsilon de PApplication de l’algorithme DBSCAN sur un jeu de données
Nous allons utiliser la bibliothèque sklearn pour faire le clustering avec DBSCAN
Import des librairies necessaires
Premièrement, nous importons les bibliothèques numpy, pyplot et sklearn .
import numpy as np
from sklearn.cluster import DBSCAN
from sklearn import metrics
from sklearn.datasets.samples_generator import make_blobs
from sklearn.preprocessing import StandardScaler
import matplotlib.pyplot as pltDepuis la librairie Scikit Learn, on a besoin de DBSCAN. On le charge depuis le sous module module cluster de sklearn.
Chargement des données
On genere un dataset.
# Generate sample data
centers = [[1, 1], [-1, -1], [1, -1]]
X, labels_true = make_blobs(n_samples=750, centers=centers, cluster_std=0.4, random_state=0)
X = StandardScaler().fit_transform(X)On peut afficher les données en exécutant chaque ligne individuellement.
#show the data
print(X)
print(labels_true)Output :
[[ 0.49426097 1.45106697]
[-1.42808099 -0.83706377]
[ 0.33855918 1.03875871]
...
[-0.05713876 -0.90926105]
[-1.16939407 0.03959692]
[ 0.26322951 -0.92649949]]
[0 1 0 2 0 1 1 2 0 0 1 1 1 2 1 0 1 1 2 2 2 2 2 2 1 1 2 0 0 2 0 1 1 0 1 0 2
0 0 2 2 1 1 1 1 1 0 2 0 1 2 2 1 1 2 2 1 0 2 1 2 2 2 2 2 0 2 2 0 0 0 2 0 0
2 1 0 1 0 2 1 1 0 0 0 0 1 2 1 2 2 0 1 0 1 0 1 1 0 0 2 1 2 0 2 2 2 2 0 0 0
1 1 1 1 0 0 1 0 1 2 1 0 0 1 2 1 0 0 2 0 2 2 2 0 1 2 2 0 1 0 2 0 0 2 2 2 2
1 0 2 1 1 2 2 2 0 1 0 1 0 1 0 2 2 1 1 2 2 1 0 1 2 2 2 1 1 2 2 0 1 2 0 0 2
0 0 1 0 1 0 1 1 2 2 0 0 1 1 2 1 2 2 2 2 0 2 0 2 2 0 2 2 2 0 0 1 1 1 2 2 2
2 1 2 2 0 0 2 0 0 0 1 0 1 1 1 2 1 1 0 1 2 2 1 2 2 1 0 0 1 1 1 0 1 0 2 0 2
0 2 2 2 1 1 0 0 1 1 0 0 2 1 2 2 1 1 2 1 2 0 2 2 0 1 2 2 0 2 2 0 0 2 0 2 0
2 1 0 0 0 1 2 1 2 2 0 2 2 0 0 2 1 1 1 1 1 0 1 1 1 1 0 0 1 1 1 0 2 0 1 2 2
0 0 2 0 2 1 0 2 0 2 0 2 2 0 1 0 1 0 2 2 1 1 1 2 0 2 0 2 1 2 2 0 1 0 1 0 0
0 0 2 0 2 0 1 0 1 2 1 1 1 0 1 1 0 2 1 0 2 2 1 1 2 2 2 1 2 1 2 0 2 1 2 1 0
1 0 1 1 0 1 2 0 1 0 0 2 1 2 2 2 2 1 0 0 0 0 1 0 2 1 0 1 2 0 0 1 0 1 1 0 2
0 2 2 2 1 1 2 0 1 0 0 1 0 1 1 2 2 1 0 1 2 2 1 1 1 1 0 0 0 2 2 1 2 1 0 0 1
2 1 0 0 2 0 1 0 2 1 0 2 2 1 0 2 0 2 1 1 0 2 0 0 1 1 1 1 0 1 0 1 0 0 2 0 1
1 2 1 1 0 1 0 2 1 0 0 1 0 1 1 2 2 1 2 2 1 2 1 1 1 1 2 0 0 0 1 2 2 0 2 0 2
1 0 1 1 0 0 1 2 1 2 2 0 2 1 1 1 2 0 0 2 0 2 2 0 2 0 1 1 1 1 0 0 0 2 1 1 1
1 2 2 2 0 2 1 1 0 0 1 0 2 1 2 1 0 2 2 0 0 1 0 0 2 0 0 0 2 0 2 0 0 1 1 0 0
1 2 2 0 0 0 0 2 1 1 1 2 1 0 0 2 2 0 1 2 0 1 2 2 1 0 0 0 1 2 0 0 0 2 2 2 0
1 1 1 1 1 0 0 2 1 2 0 1 1 1 0 2 1 1 1 2 1 2 0 2 2 1 0 0 0 1 1 2 0 0 2 2 1
2 2 2 0 2 1 2 1 1 1 2 0 2 0 2 2 0 0 2 1 2 0 2 0 0 0 1 0 2 1 2 0 1 0 0 2 0
2 1 1 2 1 0 1 2 1 2]Construction du modèle DBSCAN
Maintenant qu’on a mis les données dans le bon format (dans un Data Frame), l’entrainement de DBSCAN est facilité avec la librairie Scikit-Learn.
Il suffit d’instancier un objet de la classe DBSCAN en lui indiquant les valeurs eps et min_samples. Par la suite il faut appeler la méthode fit() pour calculer les clusters.
# Compute DBSCAN
db = DBSCAN(eps=0.3, min_samples=10)
db.fit(X)Output :
DBSCAN(algorithm='auto', eps=0.3, leaf_size=30, metric='euclidean',
metric_params=None, min_samples=10, n_jobs=None, p=None)On affiche un a un les proprietes de l’objet db pour voir ceux qu’ils contiennent.
print(db.labels_)
print(db.core_sample_indices_)Output :
[ 0 1 0 2 0 1 1 2 0 0 1 1 1 2 1 0 -1 1 1 2 2 2 2 2
1 1 2 0 0 2 0 1 1 0 1 0 2 0 0 2 2 1 1 1 1 1 0 2
0 1 2 2 1 1 2 2 1 0 2 1 2 2 2 2 2 0 2 2 0 0 0 2
0 0 2 1 -1 1 0 2 1 1 0 0 0 0 1 2 1 2 2 0 1 0 1 -1
1 1 0 0 2 1 2 0 2 2 2 2 -1 0 -1 1 1 1 1 0 0 1 0 1
2 1 0 0 1 2 1 0 0 2 0 2 2 2 0 -1 2 2 0 1 0 2 0 0
2 2 -1 2 1 -1 2 1 1 2 2 2 0 1 0 1 0 1 0 2 2 -1 1 2
2 1 0 1 2 2 2 1 1 2 2 0 1 2 0 0 2 0 0 1 0 1 0 1
1 2 2 0 0 1 1 2 1 2 2 2 2 0 2 0 2 2 0 2 2 2 0 0
1 1 1 2 2 2 2 1 2 2 0 0 2 0 0 0 1 0 1 1 1 2 1 1
0 1 2 2 1 2 2 1 0 0 1 1 1 0 1 0 2 0 2 2 2 2 2 1
1 0 0 1 1 0 0 2 1 -1 2 1 1 2 1 2 0 2 2 0 1 2 2 0
2 2 0 0 2 0 2 0 2 1 0 0 0 1 2 1 2 2 0 2 2 0 0 2
1 1 1 1 1 0 1 1 1 1 0 0 1 1 1 0 2 0 1 2 2 0 0 2
0 2 1 0 2 0 2 0 2 2 0 1 0 1 0 2 2 1 1 1 2 0 2 0
2 1 2 2 0 1 0 1 0 0 0 0 2 0 2 0 1 0 1 2 1 1 1 0
1 1 0 2 1 0 2 2 1 1 2 2 2 1 2 1 2 0 2 1 2 1 0 1
0 1 1 0 1 2 -1 1 0 0 2 1 2 2 2 2 1 0 0 0 0 1 0 2
1 0 1 2 0 0 1 0 1 1 0 -1 0 2 2 2 1 1 2 0 1 0 0 1
0 1 1 2 2 -1 0 1 2 2 1 1 1 1 0 0 0 2 2 1 2 1 0 0
1 2 1 0 0 2 0 1 0 2 1 0 2 2 1 0 0 0 2 1 1 0 2 0
0 1 1 1 1 0 1 0 1 0 0 2 0 1 1 2 1 1 0 1 0 2 1 0
0 1 0 1 1 2 2 1 2 2 1 2 1 1 1 1 2 0 0 0 1 2 2 0
2 0 2 1 0 1 1 0 0 1 2 1 2 2 0 2 1 1 1 2 0 0 2 0
2 2 0 2 0 1 1 1 1 0 0 0 2 1 1 1 1 2 2 2 0 2 1 1
0 0 1 0 2 1 2 1 0 2 2 0 0 1 0 0 2 0 0 0 2 0 2 0
0 1 1 0 0 1 2 2 0 0 0 0 2 -1 1 1 2 1 0 0 2 2 0 1
2 0 1 2 2 1 0 0 -1 -1 2 0 0 0 2 -1 2 0 1 1 1 1 1 0
0 2 1 2 0 1 1 1 0 2 1 1 -1 2 1 2 0 2 2 1 0 0 0 1
1 2 0 0 2 2 1 2 2 2 0 2 1 2 1 1 1 2 0 2 0 2 2 0
0 2 1 2 0 2 0 0 0 1 0 2 1 2 0 1 0 0 2 0 2 1 1 2
1 0 1 2 1 2]
[ 0 1 2 3 4 6 7 8 9 10 11 12 13 15 17 19 20 21
22 23 24 25 26 27 28 29 30 31 33 34 35 36 37 38 40 41
42 43 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60
61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 77 78 79
80 81 83 84 86 87 88 89 90 91 92 93 94 96 97 98 99 100
101 102 103 104 106 107 109 111 112 114 115 116 117 118 119 120 121 122
123 124 125 126 127 128 129 130 131 132 133 134 136 137 138 139 140 141
142 143 144 145 147 148 150 151 152 153 155 157 159 160 162 163 164 166
167 168 169 170 171 172 173 174 175 176 177 179 180 181 182 183 184 185
186 187 188 189 190 191 192 193 194 195 197 198 199 200 201 203 204 205
206 207 208 209 210 211 212 213 214 216 217 219 220 221 222 223 224 225
226 227 228 229 230 231 232 233 234 235 236 237 239 240 241 242 243 244
246 247 248 249 250 251 252 253 254 255 256 257 258 260 261 262 263 264
266 267 268 269 270 271 272 274 275 276 277 278 279 280 281 283 284 285
286 287 288 289 290 291 292 293 294 295 296 297 298 299 300 301 302 303
304 305 306 307 308 309 310 311 312 313 314 315 316 317 318 319 320 322
323 324 325 326 327 328 329 330 331 332 333 334 335 336 338 339 340 341
344 345 347 348 349 350 351 352 353 354 355 356 357 358 359 360 361 363
364 365 366 367 368 369 370 371 372 373 374 375 376 377 378 379 380 381
382 383 384 385 386 388 390 391 392 393 394 395 396 397 398 399 400 401
402 403 404 405 406 407 408 409 410 411 412 413 415 416 417 418 419 420
421 422 423 424 425 426 427 428 429 430 431 432 433 434 435 436 437 439
440 441 442 444 445 446 447 448 449 450 451 452 453 455 456 457 458 459
460 462 463 464 465 466 467 469 470 471 472 473 474 475 476 477 478 479
480 481 482 484 485 486 487 488 489 490 491 492 493 494 495 497 498 499
500 501 502 503 504 505 506 507 508 509 510 511 512 513 514 516 517 518
519 520 521 522 523 524 525 526 527 528 529 530 532 533 534 535 536 537
538 539 540 541 542 543 544 545 546 548 549 550 551 552 553 554 555 556
557 558 559 560 561 562 563 564 565 566 567 568 569 571 572 573 574 575
576 577 578 579 580 581 582 583 584 585 586 587 588 589 590 591 592 593
594 595 596 597 598 599 600 601 602 604 605 606 607 608 609 610 611 612
613 614 615 616 617 618 619 620 621 623 624 625 626 627 628 629 631 632
633 635 636 638 639 640 641 642 643 644 645 646 647 648 649 650 651 652
653 654 655 658 660 661 662 664 665 666 667 668 669 670 672 673 674 675
676 677 678 681 682 683 685 686 687 688 689 690 691 692 693 694 695 696
697 698 699 700 701 702 703 704 705 706 707 708 709 710 711 712 713 714
715 716 717 718 719 720 722 724 726 727 728 729 730 731 732 733 734 735
736 737 738 739 740 742 743 744 745 746 747 748 749]db.labels_ contient pour chaque element du dataset, le cluster associé . par exemple le premier et le troisieme element du dataset apartiennent au cluster « 0 », le deuxieme au cluster « 1 », le quatrieme au cluster « 2 ». les elements qui ont « -1 » sont considérer comme du bruit.
db.core_sample_indices_ contient les indices des element qui sont considérer comme noyaux. c’est à dire les point qui ont au moins 10 (min_samples) voisins qui sont à une distance inférieure ou egale à 0.3 (eps).
allons voir les metrics
core_samples_mask = np.zeros_like(db.labels_, dtype=bool)
core_samples_mask[db.core_sample_indices_] = True
labels = db.labels_
# Number of clusters in labels, ignoring noise if present.
n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0)
n_noise_ = list(labels).count(-1)
print('Estimated number of clusters: %d' % n_clusters_)
print('Estimated number of noise points: %d' % n_noise_)
print("Homogeneity: %0.3f" % metrics.homogeneity_score(labels_true, labels))
print("Completeness: %0.3f" % metrics.completeness_score(labels_true, labels))
print("V-measure: %0.3f" % metrics.v_measure_score(labels_true, labels))
print("Adjusted Rand Index: %0.3f"
% metrics.adjusted_rand_score(labels_true, labels))
print("Adjusted Mutual Information: %0.3f"
% metrics.adjusted_mutual_info_score(labels_true, labels,
average_method='arithmetic'))
print("Silhouette Coefficient: %0.3f"
% metrics.silhouette_score(X, labels))Output :
Estimated number of clusters: 3
Estimated number of noise points: 18
Homogeneity: 0.953
Completeness: 0.883
V-measure: 0.917
Adjusted Rand Index: 0.952
Adjusted Mutual Information: 0.916
Silhouette Coefficient: 0.626Visualisation des résultats du classificateur
La liste db.labels_ nous fournit à quelle cluster appartient chaque élémént. Toutefois, une liste de la sorte n’est pas très parlante. Vu que notre jeu de données est relativement petit, on peut visualiser graphiquement notre jeu de données pour observer les clusters formés.
# Black removed and is used for noise instead.
unique_labels = set(labels)
colors = [plt.cm.Spectral(each) for each in np.linspace(0, 1, len(unique_labels))]
for k, col in zip(unique_labels, colors):
if k == -1:
# Black used for noise.
col = [0, 0, 0, 1]
class_member_mask = (labels == k)
xy = X[class_member_mask & core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col),markeredgecolor='k', markersize=14)
if k == -1:
# affichage du bruit
xy_b = X[class_member_mask]
plt.plot(xy_b[:, 0], xy_b[:, 1], 'o', markerfacecolor=tuple(col),markeredgecolor='k', markersize=14)
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()Conclusion
DBSCAN présente de grands avantages par rapport aux autres algorithmes de clustering :
1 – Il n’exige pas du tout un nombre prédéfini de clusters.
2 – Il identifie les valeurs aberrantes comme des bruits différents du décalage moyen qui les jette simplement dans un cluster, même si le point de données est très différent.
3 – Il est capable de trouver assez bien des clusters de taille et de forme arbitraire.
DBSCAN présente aussi des inconvénients :
1 – Il ne fonctionne pas aussi bien que d’autres lorsque les clusters ont une densité variable (le réglage du seuil de distance ε et de minPoints pour l’identification des points de voisinage varie d’un cluster à l’autre lorsque la densité varie).
2 – Il n’est pas performant avec des données de très haute dimension car le seuil de distance ε devient difficile à estimer.
Crédit de l’image de couverture : Chire – CC BY-SA 3.0

![Spark 3 : à la découverte des RDD [Part 2]](https://penseeartificielle.fr/wp-content/uploads/2022/02/fire-1905608_1280-218x150.jpg)
![Installation de Spark 3 sous Windows [Part 1]](https://penseeartificielle.fr/wp-content/uploads/2022/02/pexels-fabiano-rodrigues-1662298-218x150.jpg)
